Na parte I deste post abordamos as funcionalidades do software Pathogen Modeling Program (PMP) Online. Hoje, falaremos do ComBase, o software de Microbiologia Preditiva mais usado mundialmente, são mais de 70.000 usuários ao redor do mundo.
O ComBase, assim como o PMP, está disponível gratuitamente. O acesso é dependente de login, mas o cadastro é bem simples e fácil de fazer. Após realizar a conexão, você poderá acessar um banco de dados com mais de 60.000 registros do comportamento microbiano em ambientes alimentares. Esse software de microbiologia foi lançado em 2003 e graças a doações tanto de dados que descrevem como os micro-organismos se comportam (crescem, sobrevivem ou morrem em meios de cultura e em alimentos), quanto monetárias ele está cada vez mais útil e funcional. Atualmente, ele é administrado pela Universidade da Tasmânia e pelo Departamento de Agricultura dos Estados Unidos – Serviço de Pesquisa Agrícola (USDA-ARS).
A primeira ferramenta disponível do software é o Browser, que realiza a pesquisa em um banco de dados da literatura científica e de diversas instituições que avaliaram o comportamento de micro-organismos deteriorantes e patogênicos em alimentos e em meios de cultura. A busca pode ser feita adicionando-se os campos: Micro-organismo (patógenos, bactérias deteriorantes e bolores e leveduras); Tipo de alimento ou meio de cultura; variadas Condições (exemplos: diferentes concentrações de conservantes, embalados à vácuo e presença de microbiota); várias Propriedades (exemplos: inoculação de esporos, cepas resistentes a antibióticos ou tolerantes a ácidos); Faixas de temperatura, de atividade de água e de pH específicas; por fim, também pode-se restringir a pesquisa por autor.
Já o Broth Models fornece previsões, a partir de modelos matemáticos, baseados em dados selecionados do banco do ComBase, em função de fatores ambientais, como temperatura, pH e atividade de água do meio de cultura. Os tipos de modelos disponíveis são de multiplicação, de inativação térmica e de sobrevivência não-térmica para diversas bactérias, todos baseados em meio de cultura. O resultado da predição, após se completar com os dados desejados de temperatura, pH e atividade de água, será em forma de gráfico e de tabela.
O Food Models permite a predição do comportamento de patógenos nos alimentos. Existem dois modelos disponíveis: Perfringens Predictor prevê a multiplicação de Clostridium perfringens durante o resfriamento de carnes e Salmonella in egg para prever a multiplicação de Salmonella resistente a antibióticos (cepa S. Typhimurium DT104) em produtos líquidos à base de ovos, entre 10 – 42°C.
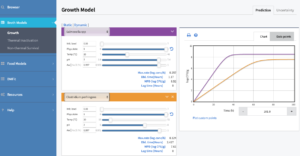
O DMFit permite que coloquemos nossos dados experimentais de contagens microbianas ao longo do tempo para ajustar esses dados aos modelos de Baranyi e Roberts, trilinear, bifásico ou linear. Após a adição dos dados experimentais, basta clicar no botão FIT e os pontos (contagem microbiana X tempo) serão ajustados aos modelos citados anteriormente. Os resultados são apresentados na forma de gráfico e de tabela. Também pode-se optar por trocar o modelo apresentado e os parâmetros serão recalculados automaticamente.
Em Resources há uma lista bastante útil de outras ferramentas de microbiologia preditiva disponíveis. Finalmente, em Help você pode aprofundar os conhecimentos e sanar dúvidas sobre todas as ferramentas do Combase citadas aqui no post, bem como assistir a tutoriais que explicam o passo a passo do uso do software.
Apesar de poder assustar um pouco no começo, essas ferramentas disponíveis gratuitamente trazem uma interface amigável à microbiologia preditiva.
Agora chega de teoria, acesse os sites do PMP e do ComBase e veja como esses softwares são fáceis de usar e acessíveis a partir de um simples clique!
Referências:
Baranyi J. and Roberts T.A. (1994). A dynamic approach to predicting bacterial growth in food. Int. J. Food Microbiol. 23, 277-294.
Baranyi J. and Tamplin M. (2004). ComBase: A Common Database on Microbial Responses to Food Environments. J. Food Prot. 67(9):1834-1840.
McMeekin J., Baranyi J., Zwietering M., Ross T., Dalgard P., Bowman J. and Kirk M. (2005). Information systems in food safety management. Int. J. Food Microbiol. 112, 181–19