Conceitos gerais e algumas aplicações da Microbiologia Preditiva foram abordadas neste post. Relembrando: a Microbiologia Preditiva expressa o comportamento microbiano – tanto de deteriorantes, quanto de patógenos – por meio de modelos matemáticos (fórmulas), levando em consideração os principais fatores para multiplicação, inativação ou sobrevivência desses micro-organismos, como, por exemplo, atividade de água, pH e temperatura.
O fato do desenvolvimento da Microbiologia Preditiva necessitar de conhecimentos em matemática, em informática e em estatística, além da própria microbiologia, acaba afastando muitas pessoas dessa incrível ferramenta. Por isso, hoje, vamos apresentar um software, disponível gratuitamente, fácil de usar e acessível a partir de um simples clique!
Estamos falando do Pathogen Modeling Program (PMP) Online, desenvolvido pelo Departamento de Agricultura e Serviço de Pesquisa Agrícola dos EUA (USDA-ARS), no início dos anos 90. Os tipos de modelos disponíveis são: multiplicação (Growth), sobrevivência (Non-Thermal Inactivation), inativação (Thermal Inactivation) e resfriamento (Cooling). Esses modelos são gerados a partir de meios de cultura e de alimentos específicos (maioria são matrizes cárneas), principalmente envolvendo o comportamento de patógenos. Em toda a previsão que será feita há uma referência dos estudos utilizados para determinar o comportamento microbiano.
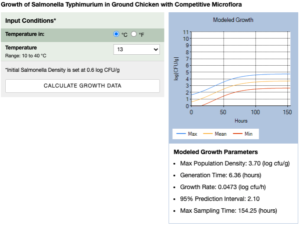
Para escolher um modelo adequado à situação para a qual você gostaria de predizer a multiplicação, a inativação ou a sobrevivência de um micro-organismo, primeiramente tente encontrar um modelo que caracterize o desenvolvimento da bactéria no alimento desejado. Considere a microbiota, como por exemplo, o modelo de Multiplicação de Salmonella Typhimurium em carne de frango moída com microbiota (Growth of Salmonella Typhimurium in Ground Chicken with Competitive Microflora). Em muitos casos, você não encontrará um modelo que corresponda exatamente à formulação do seu produto. Assim, é melhor escolher um modelo em meio de cultura (broth culture). Porém, leve em consideração que modelos desenvolvidos em meios de cultura e sem microbiota, geralmente apresentam multiplicação mais rápida dos micro-organismos, quando comparados aos modelos construídos a partir de experimentos realizados em alimentos com microbiota.
Além disso, quando for possível escolher ambientes com ou sem oxigênio, escolha um modelo anaeróbico se o seu produto for embalado a vácuo. Por outro lado, escolha um modelo aeróbio se quiser entender como as bactérias reagirão quando a embalagem for aberta e exposta ao oxigênio. Geralmente, quando as bactérias podem crescer em condições aeróbias ou anaeróbicas, o crescimento é mais rápido em condições aeróbias.
Por fim, após escolher todos os parâmetros para realizar a predição, você só vai precisar clicar no botão Calculate (calcular). O resultado aparecerá em forma de gráfico e de tabela. Também alguns modelos trazem Limites de Confiança Superior (UCL) e Inferior (LCL), que indicam a variação nas previsões com nível de confiança de 95% ou de 99%; por precaução use o UCL. Nos modelos de multiplicação, para obter uma previsão mais segura, escolha a opção “no lag”, ou seja, que não considera a fase lag (tempo necessário para a bactéria se adaptar ao ambiente e começar a se multiplicar).
Todas essas previsões, realizadas usando o PMP, são específicas para as cepas bacterianas e os ambientes usados para gerar os modelos. Assim, não se pode garantir as previsões para outras cepas e outros ambientes, sem que sejam realizados estudos de validação adequados. Além disso, é importante ressaltar que as previsões do comportamento microbiano não são 100% precisas. Variações e incertezas são introduzidas por meio de erros experimentais e ajustes dos modelos primários e secundários. Mesmo assim, a utilização da Microbiologia Preditiva é uma importante estratégia para gestão da segurança de alimentos.
Na parte II desse post apresentaremos o ComBase (https://www.combase.cc/index.php/en/), o software mais usado mundialmente!
Referências
ICMSF(1996) Microorganisms in Foods 5: Characteristics of Microbial Pathogens, Roberts, T. A., Baird-Parker, A. C. and Tompkin, R. B. (eds.), Blackie Academic & Professional, London [ISBN 0 412 47350 X]
Oscar T.P. (2006) Validation of a Tertiary Model for Predicting Variation of Salmonella Typhimurium DT104 (ATCC 700408) Growth from a Low Initial Density on Ground Chicken Breast Meat with a Competitive Microflora. Journal of Food Protection 69(9):2048-2057
Whiting, R. C. 1995. Microbiological modeling. CRC Critical Reviews in Food Science and Nutrition. 35:467-494.
US Department of Agriculture, Agricultural Research Service, Eastern Regional Research Center, Wyndmoor, Pennsylvania, USA. PMP online. www.arserrc.gov/mfs/pathogen.htm.



