As doenças transmitidas por alimentos (DTA) são causadas pela ingestão de água ou de alimentos contaminados. No Brasil, entre 2009 e 2018, foram registrados no total 6.800 surtos de DTA, envolvendo aproximadamente 120 mil doentes e 99 óbitos. O aumento dos casos de DTA está relacionado ao crescimento da população mundial, ao processo de urbanização desordenado, ao crescimento da exportação de alimentos, ao alto consumo de fast-food, entre outras questões.
Os surtos de DTA são considerados eventos de saúde pública, sendo de notificação compulsória conforme a Portaria n.º 1.061/2020 do Ministério da Saúde. No entanto, os dados registrados de casos esporádicos e surtos de DTA são considerados subnotificados por diversos motivos, como a não procura dos serviços de saúde pelos pacientes com sintomas leves e o descarte do alimento suspeito, impedindo uma investigação completa.
Os métodos tradicionais de detecção dos micro-organismos em alimentos são confiáveis, porém demandam um certo tempo para a obtenção dos resultados. A caracterização molecular é uma ferramenta útil que permite não somente identificar os micro-organismos, como também analisar o perfil clonal das cepas isoladas. A detecção mais rápida do patógeno agiliza a adoção de medidas corretivas e preventivas para evitar futuros surtos, bem como o rastreamento da fonte da contaminação.
A técnica Multilocus sequence typing (MLST) pode ser utilizada para diagnóstico e monitoramento de surtos e casos eventuais por possuir um alto nível de discriminação entre os isolados. O MLST foi idealizado em 1998 para identificar relações clonais entre bactérias. Esse método é baseado na comparação da sequência genética de sete genes constitutivos, que são expressos continuamente em condições normais de crescimento dos microrganismos. O conjunto dos genes forma um perfil alélico denominado tipo sequencial (ST, do inglês Sequence Type), que pode ser organizado em grupos relacionados formando os complexos clonais (CC) de acordo com o número de alelos compartilhados, além de definir o possível genótipo central do grupo. Essas informações são inseridas em um banco de dados online com acesso aberto que recebe, organiza, compara e disponibiliza informações sobre as sequências de ácido desoxirribonucleico (DNA) depositadas.
Baseando-se nas informações presentes no banco de dados, pode ser realizada uma comparação entre todas as cepas depositadas. Por exemplo, um isolado bacteriano de origem alimentar sofrerá o processo de extração e amplificação por reação em cadeia da polimerase (PCR) do seu DNA. Após essas etapas, realiza-se a purificação dos produtos da PCR, o sequenciamento e a análise das sequências, sendo obtido ao final um ST para o isolado. Este ST pode ser comparado com todos os outros isolados de amostras clínicas no mundo que estão depositadas no banco. Com base nas informações oriundas da análise, pode-se verificar a possibilidade de um ST isolado de determinadas categorias de alimentos estar relacionado a casos de infecção.
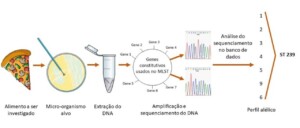
Ademais, a partir dos dados obtidos, também é possível verificar a distribuição deste ST não somente no país de origem, mas também em outros países, permitindo a realização de uma análise da distribuição geográfica e temporal. Assim, é possível obter um panorama sobre a epidemiologia e diversidade genética, auxiliando a investigação de casos esporádicos e surtos de DTA.
Autores: Paula V. Costa1, Leonardo E. O. Costa1, Janaína S. Nascimento1, Marcelo L.L. Brandão2
1Instituto Federal de Educação, Ciência e Tecnologia do Rio de Janeiro (IFRJ)
2Fundação Oswaldo Cruz, Rio de Janeiro, RJ
Referências
BRASIL. Ministério da Saúde. Manual integrado de prevenção e controle de doenças transmitidas por alimentos. Brasília: Editora do Ministério da Saúde, 2010. 158p. Disponível em:<https://bvsms.saude.gov.br/bvs/publicacoes/manual_integrado_vigilancia_doencas_alimentos.pdf> Acesso em: 24 ago. 2021.
BRASIL. Ministério da Saúde. Surtos de doenças transmitidas por alimentos no Brasil: Informe 2018. Brasília, 2019. Disponível em: <https://portalarquivos2.saude.gov.br/images/pdf/2019/fevereiro/15/Apresenta—-o-Surtos-DTA—Fevereiro-2019.pdf> Acesso em: 24 ago. 2021.
DROSINOS, E. H.; HADJILOUKA, A.; PARAMITHIOTIS, S. Molecular typing of major foodborne pathogens. In: HOLBAN, A. M.; GRUMEZESCU, A. M. Foodborne diseases, 2018. 15. ed. Academic Press, 564. cap. 13, p. 422-472.
JOLLEY, K. A; MAIDEN, M. C. Using multilocus sequence typing to study bacterial variation: prospects in the genomic era. Future Microbiology, v.9, n. 5, p. 623-630, 2014.
MAIDEN, M. C.; RENSBURG, M. J. J. V.; BRAY, J.; EARLE, S. G.; FORD, S. A.; JOLLEY, K. A.; MCCARTHY, N. D. MLST revisited: the gene-by-gene approach to bacterial genomics. Nature Reviews Microbiology, v. 11, n. 10, p. 728-736, 2013.